基本信息
- 题目:Parallel and distributed architecture of genetic algorithm on Apache Hadoop and Spark
- 作者:Hao-chun Lu,F.J.Hwang,Yao-Huei Huang
- 期刊:Applied Soft Computing Journal 95(2020) 106497
- 时间:2020.06
- 链接:[https://webvpn1.jiangnan.edu.cn/https/77726476706e69737468656265737421e7e056d234336155700b8ca891472636a6d29e640e/science/article/pii/S1568494620304361]
- 关键字:遗传算法;并行与分布式计算;Apache Hadoop;Apache Spark
摘要
- 如果遗传算子所需的迭代过程可以在并行分布式计算体系结构中实现,那么遗传算法在解决大规模优化问题方面的效率将得到提高
- 开发的计算框架以将 GA核心操作符 分配到 Apache Hadoop 中的复杂机制为特点,与云计算模型相匹配
- 所提出的体系结构可以很容易地扩展到 Apache Spark
正文
- 文章提出GAN分布式计算体系结构的并行化,是一种基于Apache的遗传算法并行分布式体系结构
- 通过使用相对高效的并行化机制调度遗传算法的核心操作符,提高了HDFS的利用率,减少HDFS作业处理的空闲时间,并且这个并行化机制可以扩展到Apache Spark上的RDD
前人设计的架构
- Verma设计的并行架构:
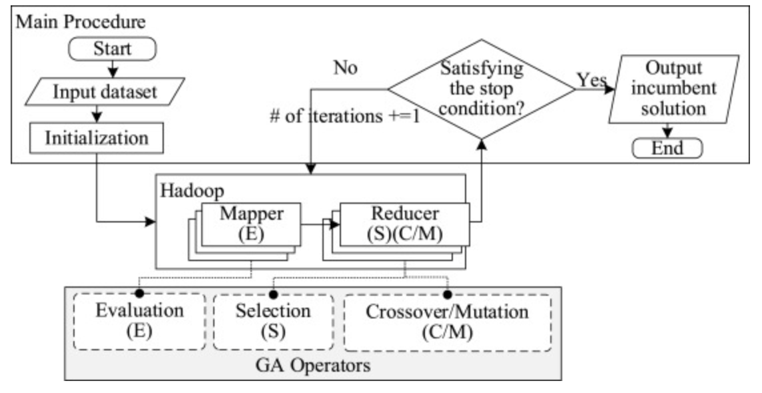
- Mapper用来评估适应度,Reducer用来选择/交叉/变异
- 缺点:
- Reducer中进化不考虑全种群,种群多样性不足会过早收敛
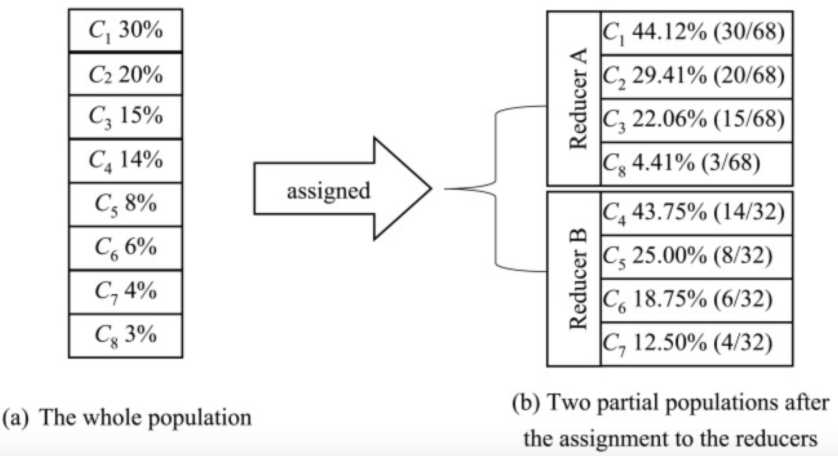
- Mapper中评估适应度后把染色体分区后传给多个Reducer,每个Reducer上的染色体GA轮盘赌概率会变化,导致选择结果不准确,如下图所示
- Kečo 和 Subasi设计的并行架构:
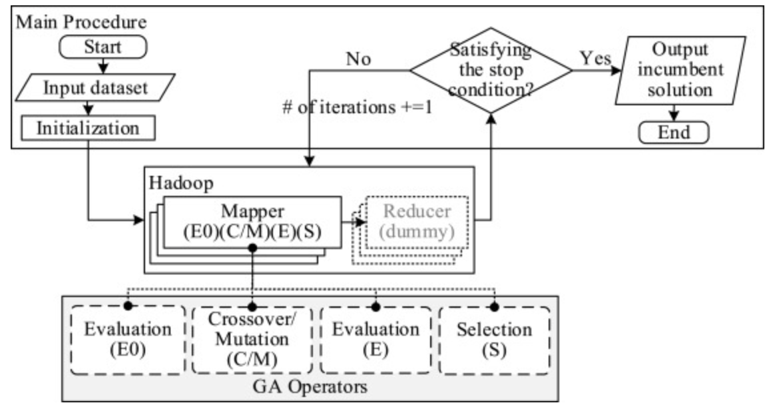
- 第一次迭代,在Mapper中首先评估初始染色体的适应度,然后交叉/变异后再评估一次,然后进行选择
- 第二次迭代开始,每次迭代不执行E0,在交叉/变异后评估适应度并判断评估结果。如果满足条件直接输出,不满足则继续迭代
- 缺点:
- 没有并行处理GA算子
本文提出的架构
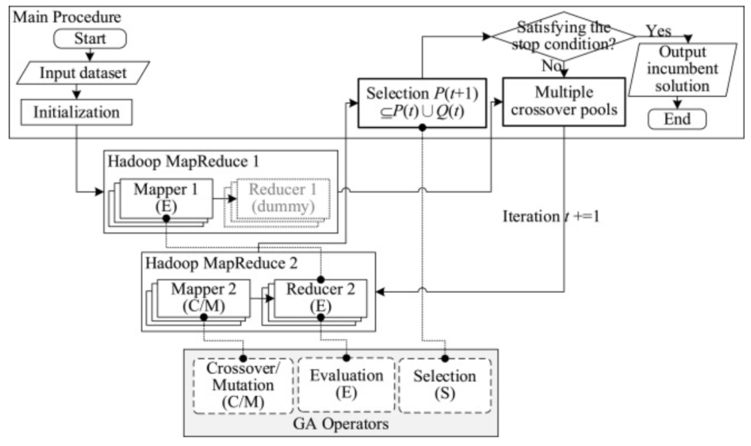
- 步骤如下:
- 首先生成初始种群,转换成子种群并激活MapReduce1
- 在MapReduce1中把每个子种群分配给多个Mapper进行评估适应度
- 激活MapReduce2,准备多个交叉池(从整个子种群中选择,组成多个交叉池,防止过早收敛)
- 迭代过程:
- 每个Mapper和Reducer对应,依次执行交叉、变异、评估,如上图所示,将结果返回到主程序
- 主程序对所有从Reducer收集到的染色体执行选择算子
- 判断停止条件,不满足则继续迭代
- 优点:
- 选择算子在主程序中
- 交叉/变异同时并行执行,产生的任何染色体都直接在Reducer中评估
将Hadoop并行架构用到Spark上
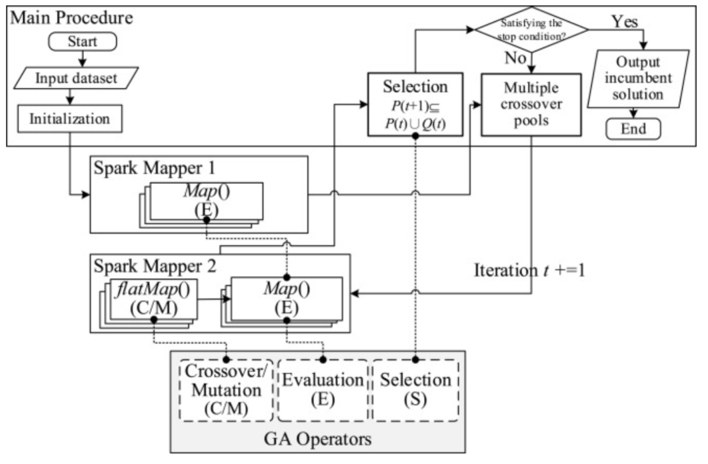
- 原理同Hadoop
结论
- 评估算子不需要其他染色体信息,独立工作,可以并行化
- 交叉算子需要在当前整个种群中选出两条染色体,不然会过早收敛。并且需要设计额外程序保证交叉后的染色体是可行解
- 变异算子调整交叉后的染色体保持多样性,也需要额外程序保证变异后是可行解
- 选择算子不能在多个子种群中进行选择,有必要把多个Reducer评估后的染色体收集起来形成一个新种群再选择,否则会导致出现Verma架构的缺点
- 设计的并行GA架构要满足上述四条
参考文献
[19] K. Gallagher, M. Sambridge, Genetic algorithms: a powerful tool for largescale nonlinear optimization problems, Comput. Geosci. 20 (7) (1994) 1229–1236.
[22] G. Luque, E. Alba, Parallel models for genetic algorithms, in: Parallel Genetic Algorithms: Theory and Real World Applications, Vol. 367, 2011.
[24] L.D. Geronimo, F. Ferrucci, A. Murolo, V. Sarro, A parallel genetic algorithm based on Hadoop MapReduce for the automatic generation of JUnit test suites, in: The IEEE 5th International Conference on Software Testing, Verification and Validation, 2012, pp. 785–793.
[26] W. Yu, W. Zhang, Study on function optimization based on master–slave structure genetic algorithm. in: The 8th International Conference on Signal Processing, 2006, pp. 3.
[27] N. Melab, E.G. Talbi, GPU-based island model for evolutionary algorithms, in: Proceedings of the 12 Annual Conference on Genetic and Evolutionary Computation, 2010, pp. 1089–1096.
[29] S. Arora, I. Chana, A survey of clustering techniques for big data analysis, in: The 5th International Conference - The Next Generation Information Technology Summit Confluence, 2014, pp. 59–65.
[30] D. Camacho, Bio-inspired clustering: basic features and future trends in the era of big data, in: 2015 IEEE 2nd International Conference on Cybernetics, CYBCONF, pp. 1–6.
[31] Y.J. Gong, W.N. Chen, Z.H. Zhan, J. Zhang, Y. Li, Q. Zhang, J.J. Li, Distributed evolutionary algorithm and their models: A survey of state-of-the-art, Appl. Soft Comput. 34 (2015) 286–300.
[33] J. Dean, S. Ghemawat, MapReduce: Simplified data processing on large clusters, Commun. ACM 51 (1) (2008) 107–113.
[37] A. Verma, X. Llorà, D.E. Goldberg, R.H. Campbell, Scaling genetic algorithms using MapReduce, in: The 9th IEEE International Conference Intelligent Systems Design and Applications, ISDA’09, 2009, pp. 13–18.
[38] D. Kečo, A. Subasi, Parallelization of genetic algorithms using Hadoop Map/Reduce, SouthEast Eur. J. Soft Comput. 1 (2) (2012).
[39] R.Z. Qi, Z.J. Wang, S.Y. Li, A parallel genetic algorithm based on spark for pairwise test suite generation, J. Comput. Sci. Tech. 31 (2) (2016) 417–427.
[42] R. Gu, X. Yang, J. Yan, Y. Sun, B. Wang, C. Yuan, Y. Huang, SHadoop: improving mapreduce performance by optimizing job execution mechanism in Hadoop clusters, J. Parallel Distrib. Comput. 74 (3) (2014) 2166–2179.


